dr hab. Anna Korzyńska, profesor IBIB - Kierownik Pracowni
prof. dr hab. Włodzimierz Klonowski ✞
mgr inż. Łukasz Roszkowiak
mgr inż. Jakub Żak
lek. med. Krzysztof Siemion
W badaniach naukowych w Pracowni korzystamy z metod analizy procesów obserwowanych/monitorowanych na mikroskopowych obrazach i ich sekwencjach dokumentujących tkanki oraz procesy życiowe komórek.
Badania są skoncentrowane na ściśle powiązanych celach:
- na rozwoju metod przetwarzania i analizy mikroskopowych obrazów barwionych cienkich skrawków tkanek i żywych komórek,
- na opracowaniu wspomagania metod diagnostycznych, terapeutycznych i badań naukowych za pomocą ilościowej analizy morfologii i zachowania indywidualnych komórek oraz ich populacji,
oraz
- na liniowej i nieliniowej analizie sygnałów oraz obrazów.
Wspomaganie mikroskopii optycznej za pomocą ilościowych metod analizy obrazów umożliwia uchwycenie zależności pomiędzy zjawiskami biologicznymi, np.: (1). pomiędzy morfologią, a zachowaniem komórek (hepatocytów i fibroblastów bez i ze zmianami genetycznymi [Korzyńska i wsp., 2017]) oraz (2) warunkami efektywnego namnażania komórek w kulturach komórkowych, (neuralnych komórek macierzystych [Korzyńska, 2007; Korzyńska i wsp., 2008]).
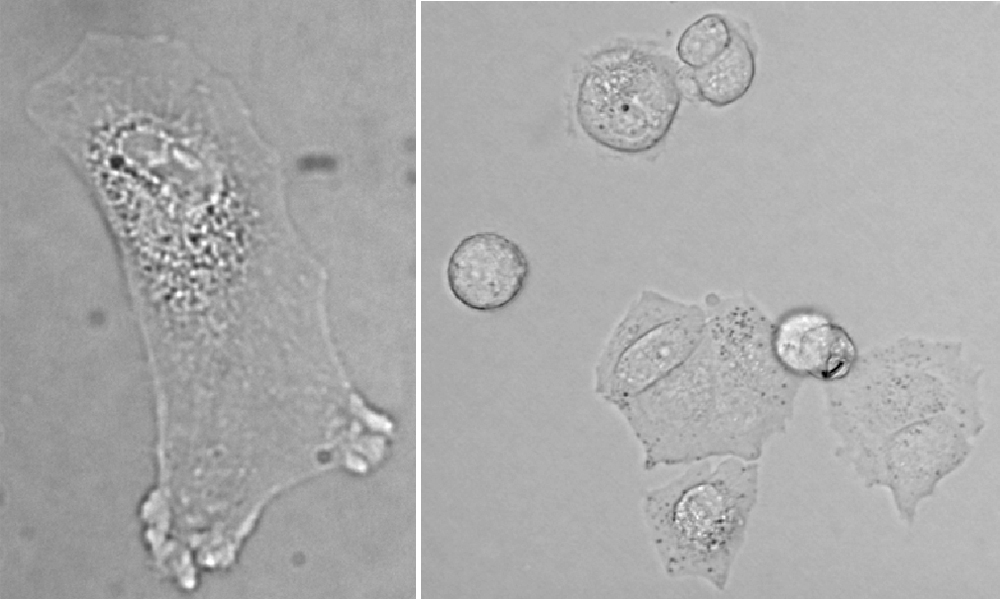
Rys. 1. Obrazy komórek obserwowanych w jasnym polu: lewy - fibroblast izolowany z skóry człowieka i prawy – neuralne komórki macierzyste linii HUCB−NSC w hodowli.
Analiza obrazów mikroskopowych tkanek barwionych w celach diagnostycznych i prognostycznych, ze wspomaganiem tego procesu za pomocą metod analizy obrazów, jest już od dawne wcielana w życie, ale wciąż nie daje zadowalających rezultatów. Wydaje się, że w dobie cyfryzacji służby zdrowia, kiedy preparaty na szkiełkach mają być zastąpione ich cyfrowymi wersjami, zwanymi wirtualnymi slajdami, lokalne cechy preparatu mogą i powinny być oceniane automatycznie przy pomocy metod analizy obrazów. W Pracowni zaproponowano ilościowe metody analizy jąder komórkowych, widocznych na obrazach cienkich skrawków tkanek pobranych w biopsji i barwionych immunohistochemicznie z użyciem DAB i hematoksyliny. Jak widać na przedstawionym przeglądzie preparatów, charakterystyka obrazów w różnych chorobach (w chłoniakach, oponiakach i raku sutka) i dla różnych przeciwciał (FoxP3, Ki67), a nawet dla różnych organów objętych tą samą chorobą (skóra i migdałki w DLBCL) jest bardzo różna i dlatego wymaga specjalizowanych metod, uwzględniających specyfikę architektury tkanki i jej zmian chorobowych.
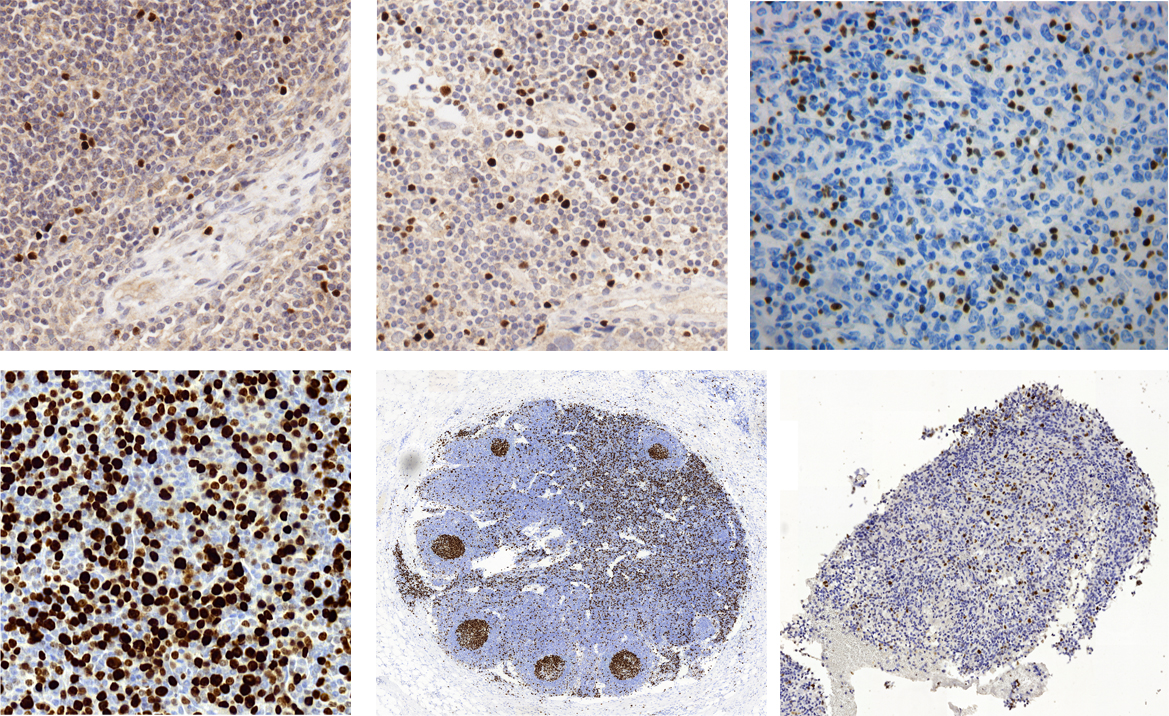
Rys. 2a. Obrazy fragmentów preparatów barwionych immunohistochemicznie z użyciem DAB&H:
- w górnym wierszu - obrazy barwione markerem przeciw FoxP3, wskazującym jądra komórek T-regulatorowych w następujących tkankach (od prawej): sutek w raku sutka, węzeł limfatyczny po przerzutach, węzeł limfatyczny w chłoniaku rozlanym i
- w dolnym wierszu - obrazy barwione przeciw markerowi Ki67 (marker proliferacji) w następujących tkankach (od prawej): skóra w rozlanym chłoniaku z dużych komórek B (ang. DLBCL), zdrowy węzeł limfatyczny, mózg w przebiegu oponiaka. (ang. meningoma)
W niektórych chorobach diagnozę stawia się na podstawie oszacowania liczby specyficznych komórek, uwidocznionych przez zastosowanie barwienia tkanek (np. eozyną i hematoksyliną lub barwienie immunohistochemiczne) oraz na postawie oceny ich architektury tkankowej.
Spośród opisanych w literaturze metod segmentacji obrazów tkanek barwionych, żadna nie jest uniwersalna i nie da się przy jej pomocy dowolnie podzielić obrazu na segmenty zawierające obiekty immunopozytywne (brązowe jądra komórkowe na prezentowanych obrazach) i immunonegatywne (niebieskie jądra komórkowe), zgodnie z percepcją specjalisty patologa. Dlatego w Pracowni są rozwijane poniżej omówione nowe metody segmentacji i udoskonalane już istniejące [Korzyńska i Zduńczuk, 2008] na drodze dostosowywania ich działania do wykorzystania charakterystycznych własności poszczególnych technik mikroskopowych [Korzyńska i Iwanowski 2008; Iwanowski i wsp., 2009] oraz wiedzy o obserwowanych komórkach i tkankach [Iwanowski i wsp., 2010, Callau i wsp., 2014].
Ponadto preparty tkankowe mogą być analizowane na podstawie biopsji tkanek stanowiących ich cienkie skrawki pochdzące od pacjenta lub jako macierze tkanek pochodzące od różnych pacjentów i barwionych w jednych warunkach. Niezależnie od sposobu przygotowania preparaty zawierają zarówno fragmenty istotne jaki i nieistotne lub wręcz stanowiące artefakty, które należy oddzielić od fragmentów nadajacych się do analizy ilościowej.
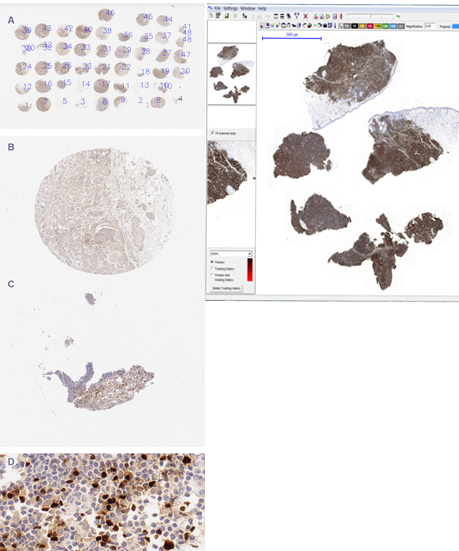
Rys. 2b. TMA i WSI
Dorobkiem Pracowni jest zaproponowanie następujących algorytmów do wykrywania i zliczania jąder komórkowych z pozytywną immunoreaktywnością, czyli zabarwionych na brązowo:
- Metoda powstała w ramach współpracy z Molecular Biology and Research Section of the Hospital de Tortosa Verge de la Cinta i z Pathology Department Hospital de Tortosa Verge de la Cinta, finansowana przez European Cooperation in Science and Technology (COST) Action, a dokładniej przez Telepathology Network in Europe 2007-2011 (EURO-TELEPATH) o symbolu IC0604 i tytule „Prospectives on Digital Pathology”, opublikowana w 2010 [Korzynska i wsp., 2010, Korzynska i wsp., 2013]. Metoda jest przeznaczona do analizy komórek znakowanych przeciwciałem FOXP3 w preparatach pacjentów z chłoniakami rozlanymi (ang. Follicular Lymphoma);
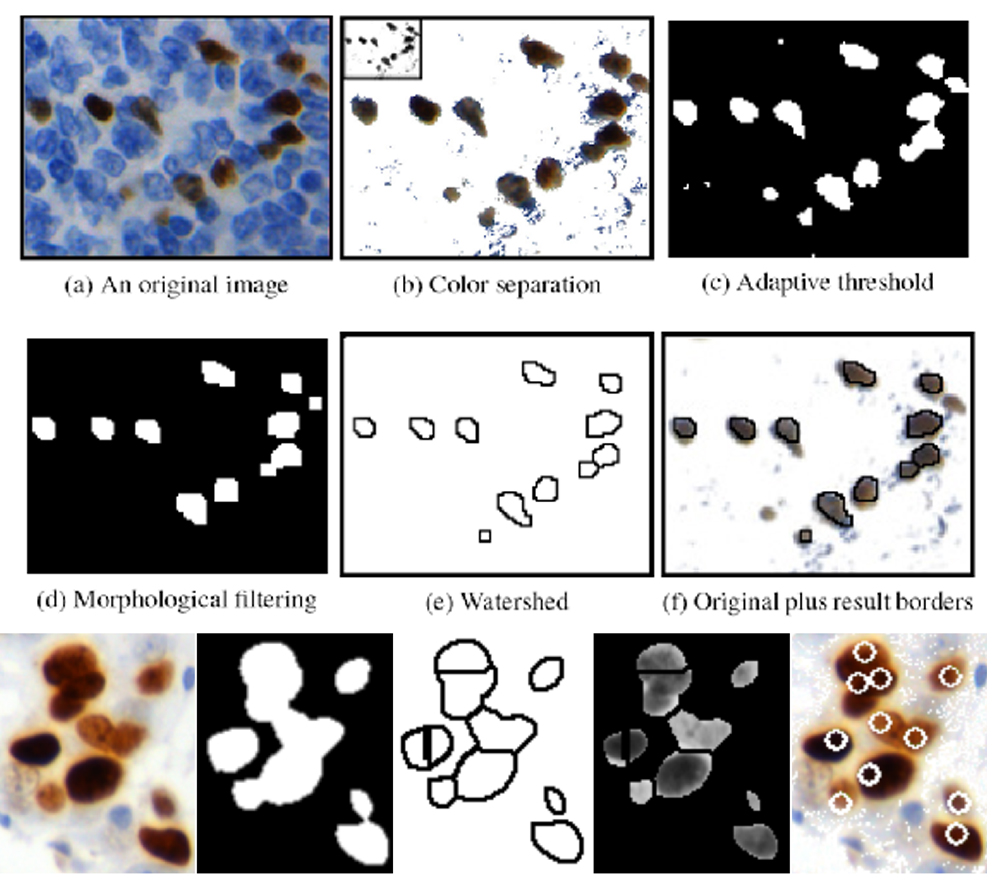
Rys. 3. Metoda zliczania ilości immunioreaktywnych jąder komórkowych oparta na separacji koloru,
progowaniu adaptacyjnym i wododziale
- Metoda METINUS [Roszkowiak i wsp., 2015a, Roszkowiak i wsp., 2015b, Roszkowiak i wsp., 2016], powstała w ramach realizacji współpracy z tym samym ośrodkiem w roku 2014, a jej modyfikacja z 2015, zwana METINUS Plus, [Korzynska i wsp., 2017] została opracowana na potrzeby projektu pt.: „Rozwój zautomatyzowanych metod ilościowego oznaczania markerów immunohistochemicznych do określenia wzorców odpowiedzi immunologicznej w raku sutka” i dotyczy analizy liczby limfocytów T-regulatorowych w preparatach pacjentek z rakiem sutka w dwóch grupach – (1) z lub (2) bez przerzutów do węzłów chłonnych.

Rys. 4. Metody Metinus i MetinusPlus: wykorzystujące parametry rozkładu jasnści jąder immunoraktywnych
i zmodyfikowaną wersję wododziału przedstawioną na schemacie po prawej stronie
- Metody MetPiKi67 z 2015 roku [plakat] na potrzeby projektu „Platforma internetowa komputerowej analizy obrazów mikroskopowych do wspomagania współczesnej diagnostyki patomorfologicznej” oraz jej modyfikacja MetPiKi67bis z 2017, wykonana w temacie statutowym [jeszcze nieopublikowana] pozwalają zanalizować obrazy barwionych skrawków tkanek od chorych z chłoniakami typu DLBCL (ang. Diffuse Large B Cell Lymphoma). W przypadku tej metody analiza ilościowa dotyczy zarówno jąder komórek immunoreaktywnych (brązowych) jak i komórek immunonedatywnych (niebieskich).
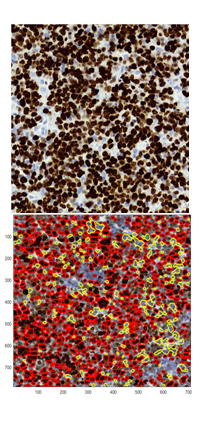
Rys. 5. Rezultaty działania metody MetPiKI67
Wszystkie metody posłużyły współpracującym z nami lekarzom do przeprowadzenia badań nad określeniem wzorca odpowiedzi immunologicznej w raku sutka [López i wsp., 2014; Callau i wsp., 2014] oraz nad analizą mikrośrodowiska potrójnie negatywnego raka sutka, który nie wykazuje pełnej odpowiedzi na chemioterapię [Lejeune i wsp., 2013].
Poza tym, na potrzeby analizy struktur histologicznych i preparatów cytologicznych, przebadaliśmy problemy standaryzacji [Korzyńska i wsp., 2010] i oceny jakości obrazu oraz zaproponowaliśmy metodę korekcji koloru na podstawie wczytania szkiełka kalibracyjnego [Korzynska i wsp., 2016, Markiewicz i wsp., 2016].

Rys. 6. Schemat korekcji koloru bazyjący na specjanie przygotowanych szkiwłkach kalibracyjnych
Zaproponowano również metodę i oprogramowanie do dzielenia obrazu wielkoobszarowego, przedstawiającego tzw. mikromacierz (ang. TMA), czyli obraz próbek wielu pacjentów zebrany na szkiełku i wspólnie barwiony. Zaproponowane oprogramowanie pozwala podzielić wielkoobszarowy obraz na poszczególne próbki i opisać je zgodnie ze schematem [Roszkowiak i wsp., 2016].
Rys. 7. Schemat działania metody PATMA
W celu oceny jakości proponowanych metod analizy ilościowej i segmentacji przygotowano narzędzia do tworzenia syntetycznych obrazów mikroskopowych o wymaganych parametrach [Iwanowski i wsp., 2009, Iwanowski i wsp., 2015a, Korzynska i wsp., 2013]. Oprogramowanie oparto na rozbudowie metody SIMPl, zaproponowanej przez Lechmusollę i współpracowników [Lehmussola i wsp., Computational framework for simulating fluorescence microscope images with cell populations. Med Imaging, IEEE Trans. 2007, 26 (7): 1010-1016 i [Lehmussola i wsp., Synthetic images of high-throughput microscopy for validation of image analysis methods. Proc IEEE. 2008, 96 (8): 1348-1360].
W Pracowni zrealizowano projekty:
- „Opracowanie metody segmentacji obrazów komórkowych na podstawie sekwencji obrazów mikroskopowych”, finansowany przez Ministerstwo Nauki i Szkolnictwa Wyższego RP nr NN518391134, kierowany przez dr Annę Korzyńską (10.08.2008 do 09.06.2011)
- „Rozwój zautomatyzowanych metod ilościowego oznaczania markerów immunohistochemicznych do określenia wzorców odpowiedzi immunologicznej w raku sutka”; projekt finansowany przez Institute of Health Carlos III w Hiszpanii i realizowany przez Molecular Biology and Research Section, Szpitala Verge de la Cinta w Tortozie, kierowany przez dr Carlosa Lopeza (1.01.2013 r. do 31.06.2016 r.)
- „Zautomatyzowana analiza mikrośrodowiska potrójnie negatywnego raka sutka, który nie wykazuje pełnej odpowiedzi na chemioterapię. Poszukiwanie wskaźników wznowy”; projekt finansowany przez Institute of Health Carlos III w Hiszpanii i realizowany przez Molecular Biology and Research Section, Szpitala Verge de la Cinta w Tortozie, kierowany przez dr Merylene Lejeune (1.01.2014 r. do 31.12.2016 r.)
- „Platforma internetowa komputerowej analizy obrazów mikroskopowych do wspomagania współczesnej diagnostyki patomorfologicznej” (akronim PIKAOM); projekt finansowany przez NCBiR jest wykonywany we współpracy z Wojskowym Instytutem Medycznym i Politechniką Warszawską, kierowany przez dr hab. Tomasza Markiewicza, prof. nadzw. (27.09.2013 do 30.10.2016)
Natomiast projekt pt.: ”Narzędzie wspomagania histopatologa w analizie wirtualnych slajdów tkanek pacjentów z rakiem sutka barwionych immunohistochemicznie z użyciem DAB&Hematoksyliny”, finansowany przez NCN, kierowany przez Łukasza Roszkowiaka, jest realizowany od 11.09.2014 - w realizacji.
Wyposażenie
W laboratorium wykorzystuje się mikroskop optyczny, pracujący w technice jasnego pola lub fluorescencji, z możliwością cyfrowego zapisu poklatkowego sekwencji obrazowych, ze sterowanymi z komputera przesłonami (do światła UV i do światła konwencjonalnego) i ze stolikiem mikroskopu (w zakresie osi Z mikroskopu).
Sława kluczowe: analiza obrazów, segmentacja obrazów, obrazy mikroskopowe, rejestracja poklatkowa, zachowania komórek, ruch komórek, analiza cyfrowych wielkoobszarowych obrazów, korekcja i standaryzacja skanowanych obrazów barwionych tkanek, patologia cyfrowa, wspomagana komputerowo morfometria ilościowa.




















